N6-甲基脱氧腺嘌呤(6mA)是DNA腺嘌呤6号位氮原子上的甲基化修饰,在多种生命体中具有重要的生物学调控功能(图1a)。最初,6mA被发现主要存在于原核生物基因组中,用于区分自身基因组和外源DNA,保护自身DNA不被限制性内切酶性降解。近年来,6mA逐渐被发现存在于真菌、衣藻、果蝇和哺乳动物等真核生物基因组(gDNA)和线粒体DNA(mtDNA)中,其动态水平的变化调控了早期胚胎发育及线粒体转录,提示6mA是真核生物基因组中继5-甲基胞嘧啶(5mC, DNA甲基化)之后的又一个全新的表观遗传修饰,阐明其甲基化酶和去甲基化酶的分子机制对于深入理解6mA的基本生物学功能具有重要意义。
2022年6月2日,基础医学院张良研究员团队联合上海中医药大学陈红专教授团队在Nature Chemical Biology杂志上发表了题为“A fungal dioxygenase CcTet serves as a eukaryotic 6mA demethylase on duplex DNA”的研究论文,发现真菌双加氧酶CcTet是一种真核生物dsDNA 6mA去甲基化酶。灰盖鬼伞菌(Coprinopsis cinerea)是一种常见的真菌蘑菇,其基因组上存在大量的5mC和6mA修饰,且随着其生长周期变化呈现动态分布。张良研究员前期工作中已发现灰盖鬼伞菌双加氧酶CcTet通过将基因组上5mC逐步氧化成5hmC、5fC和5caC来参与DNA 5mC去甲基化调控(JACS, 2014, 136, 4801-4804)。目前研究工作进一步揭示了CcTet识别和催化dsDNA上6mA底物的分子机制,并发展了具有6mA去甲基化活性的单功能CcTet突变体D337F,为进一步研究真核生物6mA的调控机制和功能提供了新的化学生物学工具。Nature Chemical Biology在同期还配发了专家点评文章,对该工作进行了详细介绍和评论。

通过对CcTet潜在底物进行大量筛选,发现CcTet对dsDNA上的6mA具有显著的去甲基化活性(图1b),并对C-6mA-G序列具有更高的底物偏好性,这与之前报道的多种真核生物基因组6mA motif序列吻合。同时,CcTet在体外(灰盖鬼伞菌gDNA和绿藻gDNA)和体内(大肠杆菌中异源表达)也表现出了较高的基因组6mA去甲基化活性。
为阐明CcTET的6mA去甲基化分子机制,进一步解析了CcTet及CcTet与含有6mA修饰的短链dsDNA的复合物晶体结构,揭示CcTet通过3个柔性Loop区域特异识别dsDNA,并利用活性袋中两个关键氨基酸Gly331和Asp337选择性识别和催化6mA碱基的分子机制(图1c)。同时,通过与人源5mC去甲基化关键双加氧酶TET2比较发现,TET2活性口袋中与CcTet Gly331对应的氨基酸Asn1387侧链阻挡了TET2对6mA底物的容纳,将TET2 Asn1387突变成无侧链的Glycine,则能使TET2获得6mA的去甲基化活性。此外,通过与RNA m6A去甲基化酶FTO比较则发现,CcTet与FTO采用了截然不同的核酸单双链识别机制,底物碱基进入活性口袋的方向也因此发生了约90度的变化,造成FTO不能催化dsDNA的6mA去甲基化。
为获得具有区分dsDNA上5mC和6mA底物的单功能6mA去甲基化酶,开展了基于结构的定向进化筛选,通过对活性口袋中关键氨基酸突变体进行活性筛选和Dot blot实验验证,发现CcTet D337F在保持6mA去甲基化活性的同时,丧失了对5mC的催化活性。因此,CcTet D337F可以被进一步开发成能特异性作用于6mA而不受其他修饰(如5mC)干扰的化学生物学新手段(图1d)。
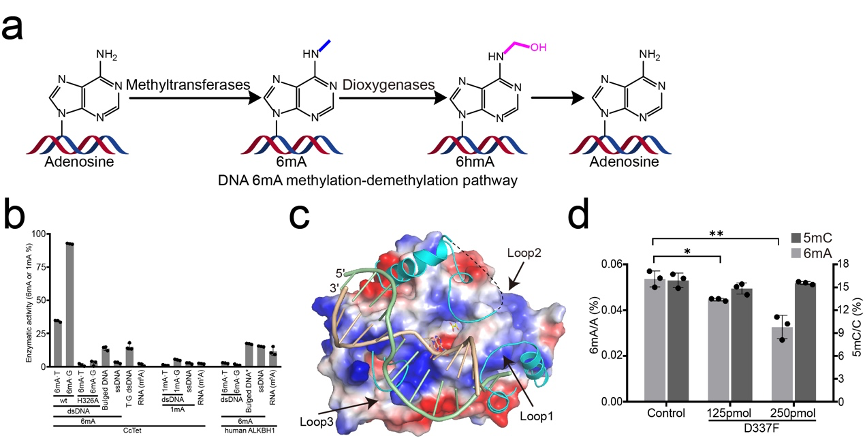
图1. dsDNA 6mA去甲基化酶CcTet的分子机制
上海交通大学医学院药理学和化学生物学系张良研究员、上海中医药大学上海市中药化学生物学前沿科学基地陈红专教授和上海交通大学医学院博士后张琳为该论文的共同通讯作者。上海交通大学医学院20级博士生穆雅娟、博士后张琳和21级博士生胡静燕为该论文的共同第一作者。该研究工作受到芝加哥大学何川教授、上海交通大学医学院附属仁济医院林厚文教授和南京师范大学刘中华教授等指导和大力支持。研究工作获得国家自然科学基金委“生物大分子动态修饰与化学干预”重大研究计划、国家自然科学基金委优秀青年基金和上海市教委曙光计划等项目资助。
原文链接:https://www.nature.com/articles/s41589-022-01041-3