近日,上海交通大学公共卫生学院、上海交通大学医学院单细胞组学与疾病研究中心郑小琪课题组在基因组学权威杂志GenomeResearch上发表题为STCC enhances spatial domain detection through consensus clustering of spatial transcriptomics data的研究论文。该研究创新性地开发了针对空间转录组数据设计的集成聚类框架STCC(Spatial Transcriptomics Consensus Clustering),显著提升了空间域检测的性能。

随着空间转录组技术(Spatially Resolved Transcriptomics, SRT)的快速发展,研究人员已能够在保留细胞空间位置的同时获取其基因表达谱信息,为揭示组织发育和疾病机制提供了革命性研究工具。然而,当前空间转录组研究面临关键瓶颈问题:作为基础分析的关键步骤,空间域检测(spatial domain detection)算法的性能在不同数据集和测序平台间存在显著差异,由此带来的不稳定性严重影响了空间异质性分析、细胞互作研究等后续分析步骤。传统单细胞RNA测序领域的集成聚类方法(如SC3)虽在批量转录组和单细胞数据中表现优异,但其在空间转录组数据中的适用性尚未得到验证。
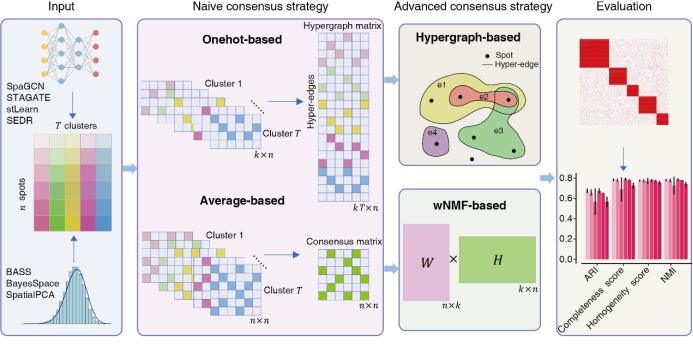
图1 STCC模型架构
本项研究提出了一种专为空间转录组(SRT)数据聚类设计的集成框架——STCC(Spatial Transcriptome Consensus Clustering)。STCC通过构建超图矩阵或共识矩阵,有效整合来自多种基线聚类算法的结果,提升空间域识别的鲁棒性与准确性。该框架共实现了四种集成策略,包括两种朴素策略(独热编码集成和平均集成)和两种高级策略(超图集成和加权非负矩阵分解集成),后者引入了超图划分、非负矩阵分解和二次规划等算法以获取更稳健的集成标签(图1)。为全面验证STCC的性能,作者在涵盖不同测序平台、物种及组织类型的七个真实数据集上进行评估,结果表明,STCC在多种聚类评价指标下均展现出卓越的整合能力和广泛的适应性。
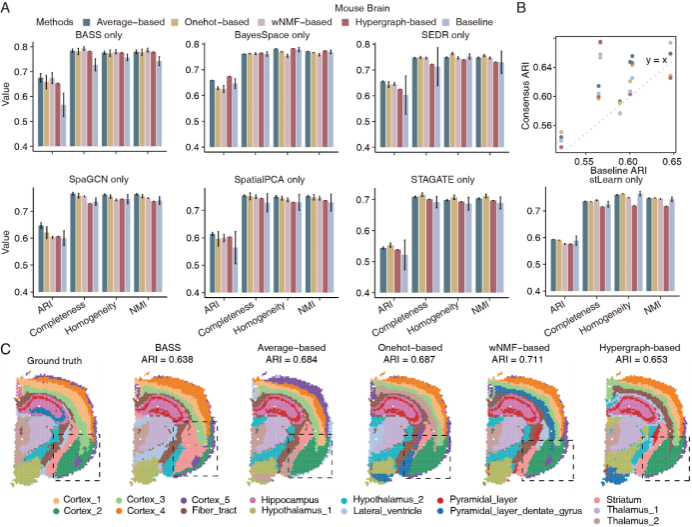
图2 STCC集成策略在单一聚类算法上的表现
研究人员首先探讨了STCC在仅使用单一基线聚类算法作为输入时的表现。通过将四种集成策略应用于七种主流空间域检测算法(如SEDR、SpatialPCA、BayesSpace等)在真实鼠脑数据上的聚类结果,发现所有STCC策略在大多数评估指标中均优于原始基线方法。尤其在“SEDR only”与“stLearn only”的情境下,STCC展现出更低的性能波动,显著增强了聚类结果的稳定性。此外,在鼠脑数据上,STCC策略成功纠正了基线算法错误识别的皮质区域结构,其中基于加权非负矩阵分解的集成策略更是精准重构了鼠脑的Cortex_5区域,与人工标注高度一致(图2)。

图3 STCC集成策略准确性和稳定性的综合评估
STCC集成框架在整合多个基线聚类算法时表现出显著优势。通过对四个真实空间转录组数据集的系统评估,研究人员发现:基线算法本身的质量显著影响共识聚类的准确性,基线算法性能越高,最终整合效果越好。例如,在小鼠嗅球数据中,不同基线算法整合得到的聚类精度差异高达0.57。进一步分析表明,对于组织结构清晰、分层明显的样本(如鼠脑、人脑背外侧前额叶皮层等),随着被整合的基线算法数量增加,集成聚类表现稳步提升。然而,对于细胞类型分布较分散的癌症样本数据,单一算法集成即可获得较优效果。在多个评估指标中,平均集成和超图集成在准确性与稳定性方面均领先,能更稳健地识别不同类型细胞,尤其是在稀有细胞群体中优势明显(图3)。这一发现表明了根据数据特征灵活选择集成策略与基线算法组合的重要性,为未来空间转录组数据分析提供了实用参考。
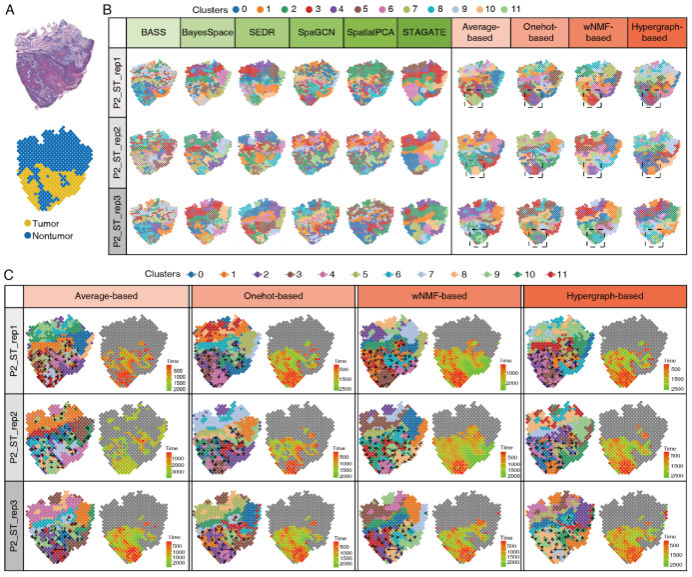
图4 STCC集成策略在鳞状细胞癌数据数据的探索性分析
在一项针对人类鳞状细胞癌(SCC)的数据分析中,STCC集成聚类框架再次展示出强大能力。研究团队使用ST技术获取的12张SCC组织切片数据,选取其中3张来自同一患者的切片作为代表进行分析。由于该数据集缺乏明确的空间结构标注,研究人员以病理学家划定的肿瘤与非肿瘤区域作为参考标准进行评估。与表现分散、结构模糊的六种基线算法相比,STCC四种共识策略准确识别出下方非肿瘤区域。基于集成聚类的结果,研究人员进一步构建了从非肿瘤区域向肿瘤区域的发育轨迹,发现非肿瘤细胞处于更早的“伪时间”阶段,提示其可能为肿瘤细胞的前体或邻近影响区域(图4)。这些结果不仅揭示了肿瘤区域与周边组织的潜在发育关系,也表明STCC在缺乏已知标签的复杂样本中,仍可提供可靠的结构解析和发展路径推断,为理解肿瘤发生发展机制提供了新线索。
上海交通大学医学院单细胞组学与疾病研究中心的郑小琪教授和北京大学医学部精准医疗多组学研究中心的吴华君研究员为该论文的通讯作者,上海交通大学医学院单细胞组学与疾病研究中心的胡聪聪博士为论文的第一作者。北京大学的魏娜娜博士和上海交通大学的杨纪元博士参与了本课题的数据分析和模型开发工作。该研究得到国家重点研发计划、国家自然科学基金、上海市科技创新行动计划自然科学基金等经费的支持。
原文链接:https://www.genome.org/cgi/doi/10.1101/gr.280031.124