在过去的十年中,单细胞转录组测序(scRNA-seq)技术的快速发展,使得研究人员可以在单细胞分辨率上分析整个转录组,从而加深了我们对发育和疾病的理解[1,2]。识别和表征复杂组织中的细胞类型,被广泛的应用于揭示细胞间的异质性以获得新的生物学见解[3]。其中无监督聚类方法在识别细胞类型方面发挥了重要的作用。近年来,scRNA-seq实验的规模迅速增长,已经产生了多套超百万细胞级别的数据[4-6],然而现有的单细胞聚类算法却难以高效的处理如此规模的数据集,更加难以应用在千万细胞的图谱级数据集上。
2022年12月5日,北京大学基础医学院精准医疗多组学研究中心吴华君课题组,上海交通大学公共卫生学院单细胞组学与疾病研究中心郑小琪课题组以及上海交通大学自然科学研究院刘林课题组合作在PLOS Computational Biology上发表了题为 “Secuer: ultrafast, scalable and accurate clustering of single-cell RNA-seq data”的学术论文。文中提出了一种基于谱聚类的用于单细胞测序数据的快速聚类算法Secuer(https://github.com/nanawei11/Secuer),能够在3到4分钟内完成对超大型数据集(1千万个细胞)的无监督聚类任务。

Secuer(a scalable and efficient spectral clustering algorithm)基于谱聚类算法(图1),从单细胞数据中识别锚点(anchors),然后借助近似最近邻算法(MAKNN)构造一个由细胞和锚点组成的加权二部图,其权重采用局部加权的高斯核距离度量。此外,作者引入了两种自动确定聚类个数的策略:1)基于锚点的图分割(如Louvain)算法;2)基于加权二部图拉普拉斯(Laplacian)矩阵的特征值的分布算法。此外,得益于算法运行时间上的优势,作者进一步引入了一个集成聚类的方法Secuer-consensus,通过变换不同的参数使用Secuer取得多个聚类结果,进而构造一个集成二部图获得一致性聚类结果(图2)。
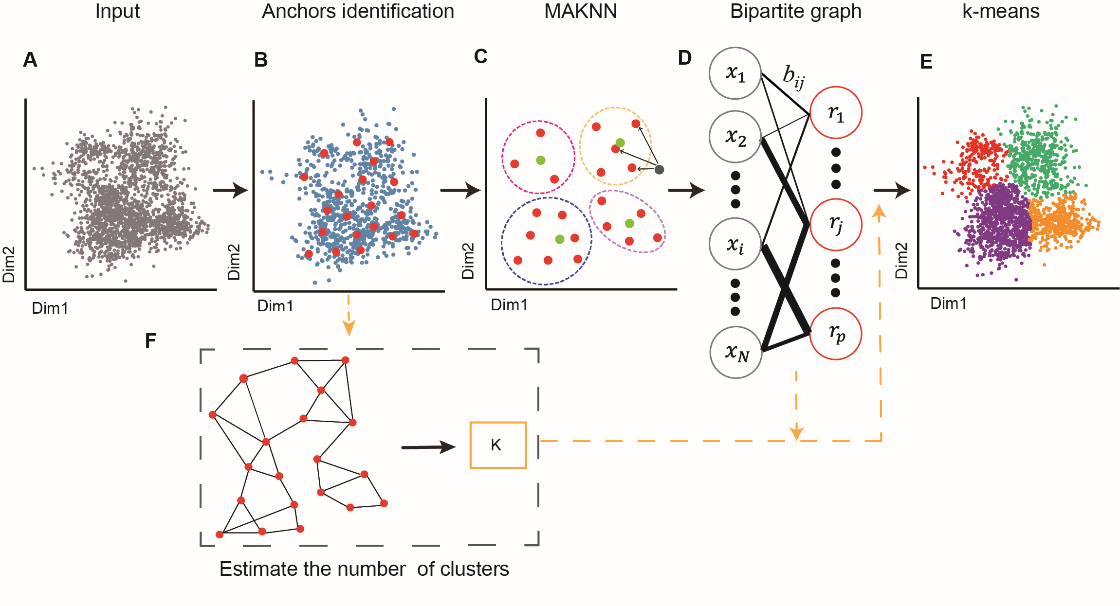
图1 Secuer流程示意图
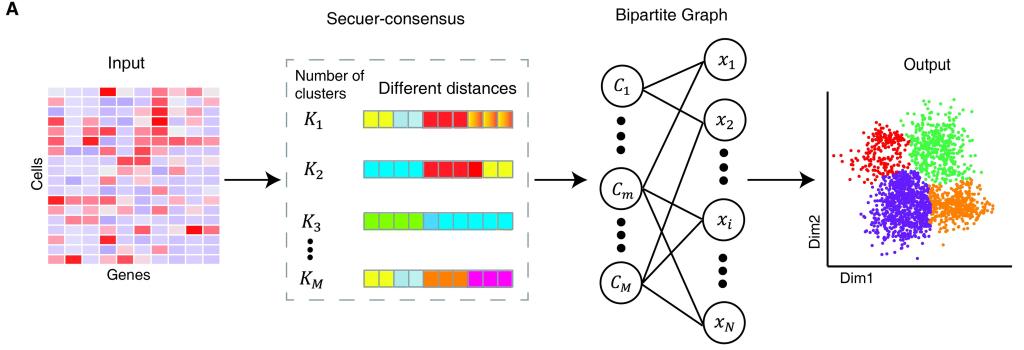
图2 Secuer-consensus流程示意图
为了证明Secuer的性能,作者首先基于小鼠大脑数据集模拟了不同数量(1万到4千万)的单细胞数据。与其他方法相比,Secuer极大的降低了聚类时间,并取得了较高的准确度。对于超大型数据集,Secuer比k-means快5倍,比Louvain/Leiden快12倍,使用的内存仅是Louvain的10% (图3)。此外,当样本量大于500万时,Secuer基于锚点空间推断的聚类个数仍然是准确的。随后,作者收集了15套涉及不同测序技术的具有注释的单细胞数据集,其中细胞个数从49到140万。Secuer相比其他方法产生了相当或更高的准确度,且平均能节省90%的运行时间。
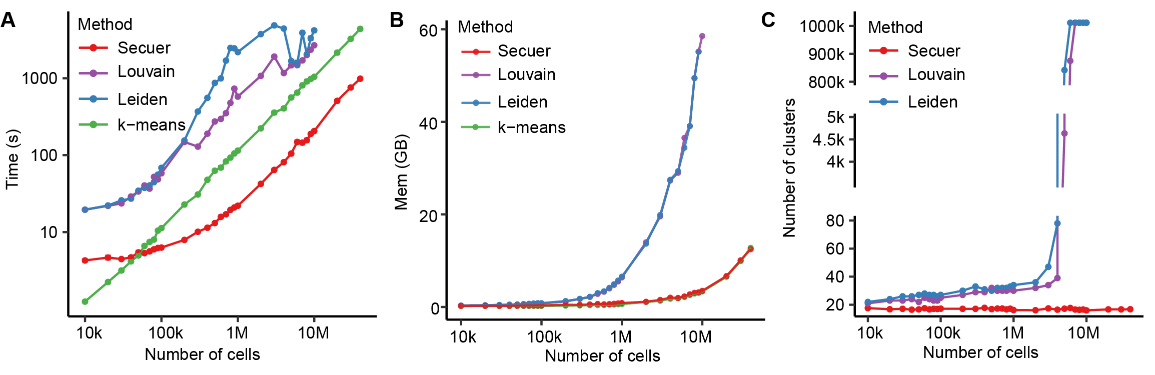
图3 模拟数据集上不同方法的结果展示
作者进一步评估了Secuer-consensus的准确度,与流行的集成聚类算法SC3相比,Secure-consensus在14个基准数据集上获得了更好的聚类精度,且速度比SC3快100倍,并且可以在SC3不能使用的大型数据集上工作。例如,Secuer-consensus可在2分钟内实现大型数据集(~140万个细胞)的聚类。与最近发表的另一个针对大规模scRNA-seq数据的集成聚类方法Specter相比,Secure-consensus在大型数据集上具有更高的准确性同时耗时更短。
总体来说,Secuer在准确性、计算成本和可扩展性之间取得了很好的平衡,是聚类图谱级scRNA-seq数据的高效算法,也可以应用于在线scRNA-seq计算平台对海量数据进行实时分析。
上海师范大学数理学院博士研究生魏娜娜为该论文的第一作者。上海交通大学数学科学学院刘林副教授、上海交通大学公共卫生学院郑小琪教授和北京大学基础医学院/北京大学肿瘤医院吴华君研究员为该论文的共同通讯作者。
原文链接:https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1010753
软件链接:https://github.com/nanawei11/Secuer
参考文献
1. Kolodziejczyk AA, Kim JK, Svensson V, Marioni JC, Teichmann SA. The technology and biology of single-cell RNA sequencing. Mol Cell. 2015;58(4):610-20.
2. Ziegenhain C, Vieth B, Parekh S, Reinius B, Guillaumet-Adkins A, Smets M, et al. Comparative analysis of single-cell RNA sequencing methods. Mol cell. 2017;65(4):631-43. e4.
3. Wang D, Bodovitz S. Single cell analysis: the new frontier in ‘omics’. Trends Biotechnol. 2010;28(6):281-90.
4. Brbić M, Zitnik M, Wang S, Pisco AO, Altman RB, Darmanis S, et al. MARS: discovering novel cell types across heterogeneous single-cell experiments. Nat Methods. 2020;17(12):1200-6.
5. Ren X, Wen W, Fan X, Hou W, Su B, Cai P, et al. COVID-19 immune features revealed by a large-scale single-cell transcriptome atlas. Cell. 2021;184(7):1895-913. e19.
6. Duò A, Robinson MD, Soneson C. A systematic performance evaluation of clustering methods for single-cell RNA-seq data. F1000Res. 2018;7.